projet BioloGrist
Optimiser la solution Grist pour la gestion des données biologiques, de la saisie des données de terrain à la soumission des données de séquençage de génomes dans les banques publiques
Responsables
Alexis DEREEPER (UMR PHIM, IRD) courriel
Juliette HAYER (UMR MIVEGEC, IRD) courriel
Résumé
A Montpellier, les unités de recherche PHIM et MIVEGEC expérimentent l’utilisation de la solution de base de données Grist pour faciliter la gestion de données de recherche. Une instance Grist a été mise en place au sein de l’équipe XPLAIN afin de centraliser et d’assurer la traçabilité des données relatives aux collections de phytopathogènes bactériens du genre Xanthomonas : des données d’échantillonnage sur le terrain (localisation GPS, dates de collecte, photos d’échantillon) jusqu’à la soumission des données de séquençage de génomes de microbes dans les banques publiques. Cette solution est envisagée dans l’unité PHIM pour l’étendre à d’autres pathogènes (champignons notamment) dans un but de gestion de collection de pathogènes mais aussi plus largement pour le suivi épidémiologique de maladies des plantes.
Dans la même optique, l’unité MIVEGEC expérimente également cette solution Grist via l’instance gouvernementale pour la gestion des souches microbiennes collectées sur les patients, leurs animaux et leur environnement, en vue de leur séquençage, notamment pour l’étude des mécanismes de résistance aux antibiotiques et de sa transmission.
Le projet proposé vise à mutualiser les efforts et consolider les bases de cette solution pour permettre de disposer d’un système souple (i) facilitant la saisie de données par les membres des équipes de recherche et leurs partenaires, (ii) permettant la mise à disposition de tableaux de bord synthétiques des données, et (iii) assurant la traçabilité des données et apportant les bases d’un plan de gestion de données (PGD) de séquençage.
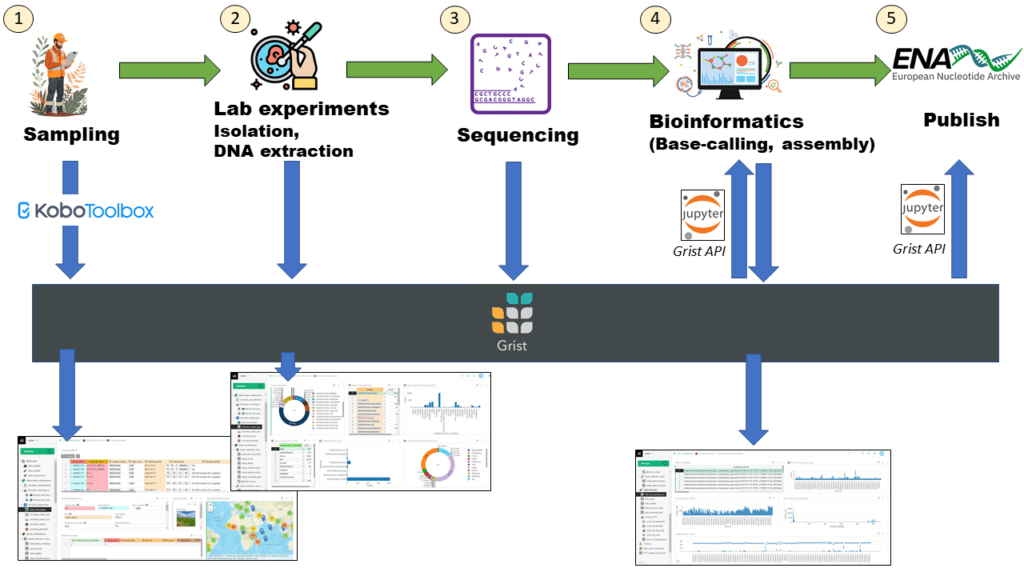
Schéma illustrant le projet BioloGrist